应用方向 调查方向
单核苷酸变异和定相
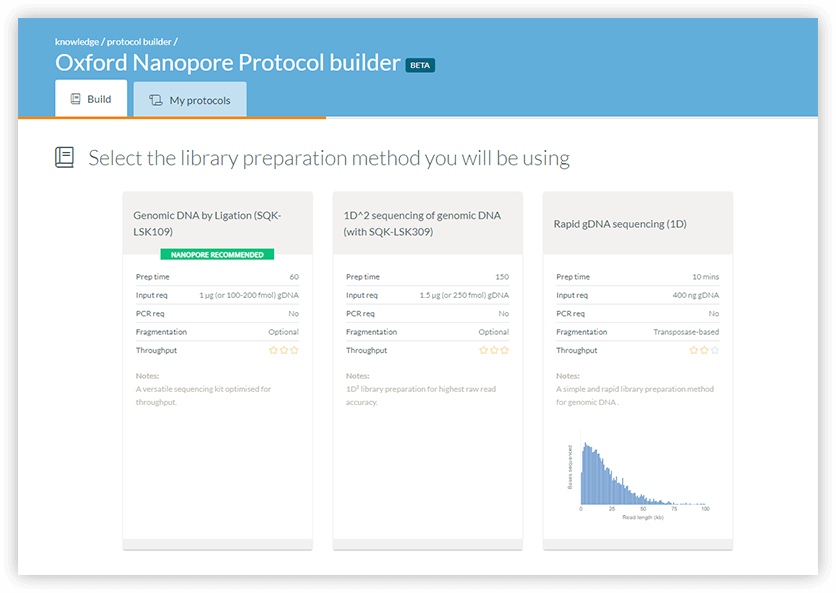
单核苷酸变异 (SNV) 与表型变异及疾病的相关性已得到了广泛研究,它们也被用于对单倍型进行定相(phasing)。然而,传统测序技术对 PCR 的需求使得 SNV 检测仅限于扩增区域,并且短读长也令解析单倍型分型具有挑战。在使用长读长纳米孔测序时,无需进行 PCR,使其能够揭示其他技术无法探及的区域中的单核苷酸多态性(SNP),并大幅简化定相。

使用长测序读长对 SNV 进行定相,并解析复合杂合性

以高精度(precision)和查全率(recall)进行实时分析

据您的要求使用一系列纳米孔测序平台根调整规模