应用方向 调查方向
融合转录本
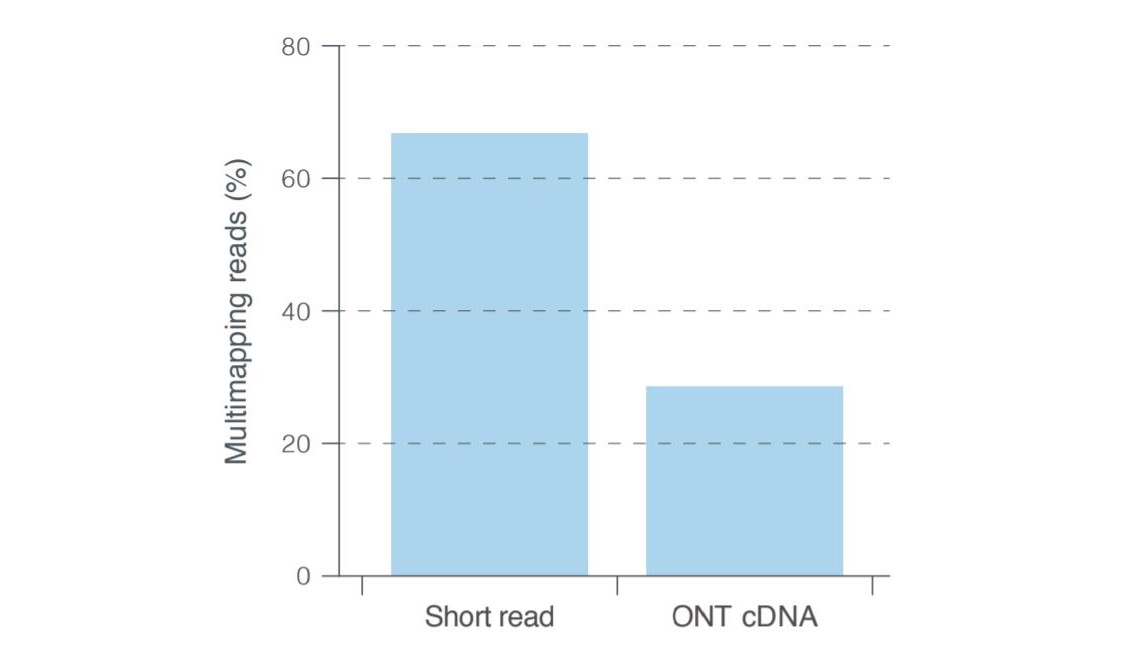
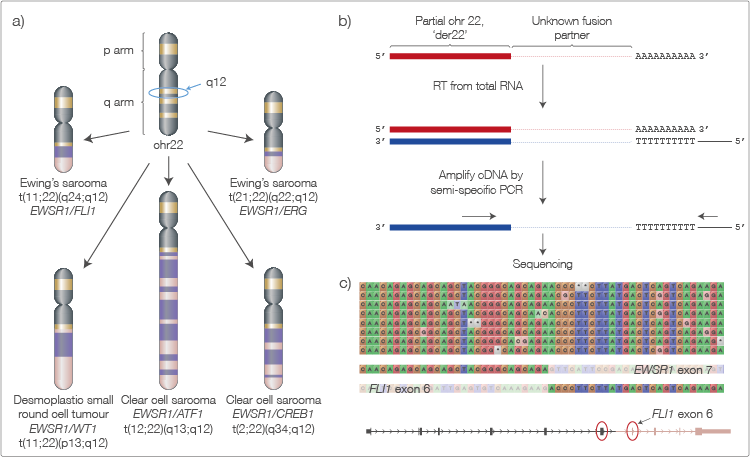
准确鉴定融合转录本对疾病(包括某些类型的癌症)的临床研究十分重要。但通过传统短读长测序方法进行识别时,需要以小片段形式对转录本进行测序,然后再进行计算组装,从而可能导致多重定位和错误组装。使用长读长纳米孔测序时,可以在单个读长中对融合转录本进行端对端测序,从而对融合及其精确的剪接连接进行综合鉴定。

使用纳米孔长读长测序全长融合转录本

通过简单、灵活的工作流程标靶融合转录本,含 PCR 或无需 PCR

通过实时测序和分析快速识别融合

使用纳米孔长读长测序全长融合转录本

通过简单、灵活的工作流程标靶融合转录本,含 PCR 或无需 PCR

通过实时测序和分析快速识别融合
“Nanopore 测序完全符合临床融合检测的需求。”William Jeck,2018 年纳米孔社区科研团体大会

如果您对我们的产品或服务有任何疑问,请直接告诉我们的销售团队成员。
联系我们您可以通过下方的二维码关注我们的技术和服务微信公众号(NanoporeSupport),与我们的技术专家交流。
