COVID-19
研究人员利用纳米孔测序技术生成了对抗 COVID-19 传播的必需数据,从 SARS-CoV-2 病毒基因组的初始表征到变体的快速鉴定。
在这里,您可以找到纳米孔测序用于基因组流行病学的信息,包括现有工作流程可以实现的结果、如何选择符合您要求的测序方法,以及开始使用的前期准备等。
本页重点介绍使用纳米孔测序研究SARS-CoV-2 病毒和 COVID-19。如需了解 Oxford Nanopore 开发的 COVID-19 诊断检测 LamPORE 的信息,请访问 Oxford Nanopore Diagnostics 网站。
了解 LamPORE COVID-19
-
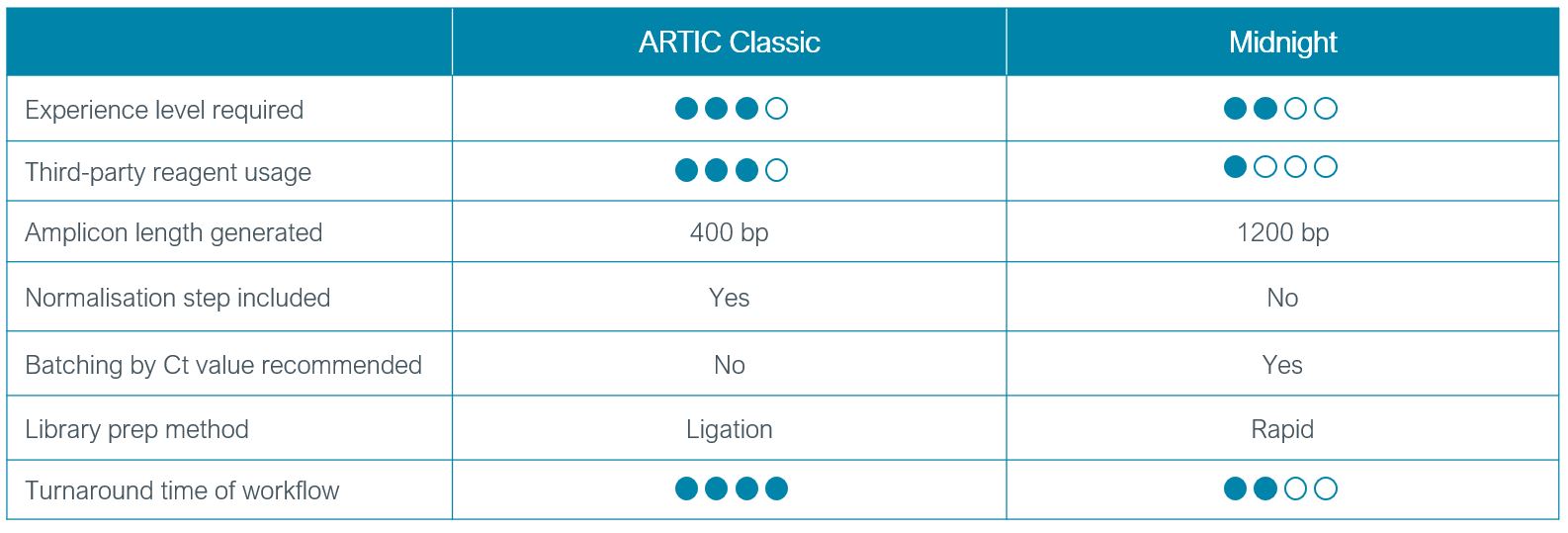
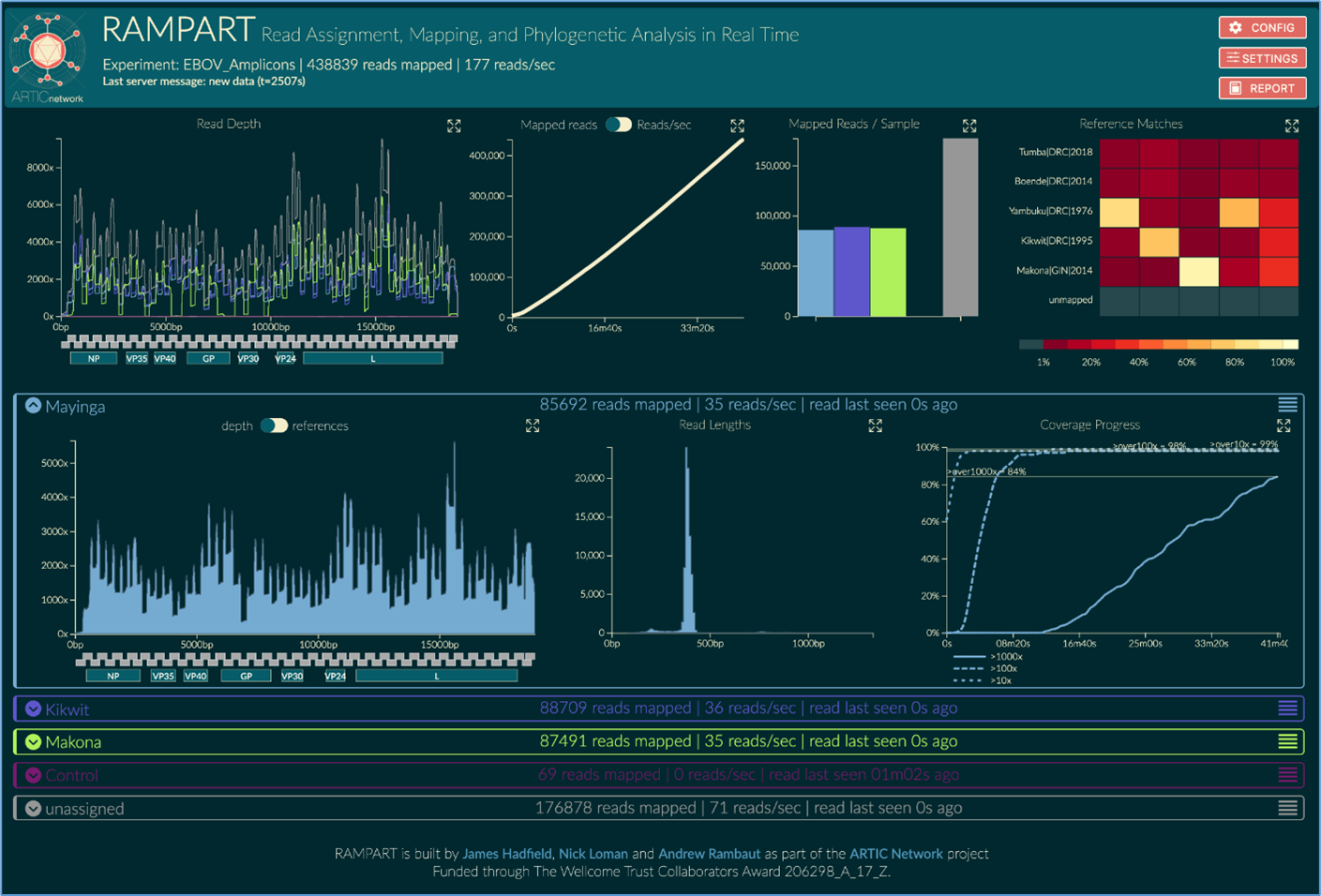
进行 SARS-CoV-2 基因组快速测序并鉴定变异
-
从便携式、分散式测序到高通量、自动化工作流程,根据您的需求调整规模
-
快速安装调试,几天之内即可使用测序技术,无需资本成本